PNU 리서치
- 메인으로 이동
- 연구/산학
- PNU 리서치

부산대 국제공동연구팀이 사람 눈의 망막처럼 빛을 보자마자 신호를 스스로 키우고 기억하는 새로운 이미지 센서를 개발했다. 인간 망막의 신경 신호 전달 방식을 모사해 빛을 감지함과 동시에 신호를 증폭하고 기억하는 방식으로, 향후 인공지능 카메라와 자율주행·로봇 비전, 인공 시각 보조 기술 등 다양한 활용이 기대된다.
전기전자공학부 심현석(사진) 교수 국제공동연구팀은 인간의 시각 경로(망막-뇌)처럼 빛을 ‘보고-처리하고-기억하는’ 기능을 하나의 구조에서 동시에 수행하는 차세대 곡면형 뉴로모픽(Neuromorphic·뇌신경모사형) 이미징 소자를 개발했다. 이번 성과는 기존 카메라 센서가 촬영(감지)과 처리·기억이 분리돼 전력 소모와 데이터 병목이 컸던 한계를 극복하고, 미래 저전력·고효율 인공지능 비전 시스템 구현을 앞당길 핵심 기술로 주목된다.
연구팀은 실리콘 태양전지(silicon solar cell)와 전해질 기반 시냅스 트랜지스터를 직렬로 연결한 캐스케이드(cascade) 광-전(optical-electrical) 시냅스 구조를 통해, 인간 망막에서 나타나는 시냅스 촉진(synaptic facilitation) 특성을 높은 수준으로 구현했다. 이 구조를 다수 집적하고, 키리가미(kirigami, 절개·접기) 구조 기반 설계를 적용해 사람 눈과 유사한 곡률의 곡면 뉴로모픽 이미저를 제작함으로써, 기존 이미지 센서와 달리 빛 감지와 동시에 신호 전처리 및 기억 기능 수행이 가능함을 실험으로 입증했다.
일반적인 이미지 센서는 빛을 전기 신호로 변환하는 역할에 주로 머물며, 인식·판단 등 고차 처리는 별도의 인공지능 프로세서가 담당한다. 이 과정에서 데이터 이동이 과도해져 병목 현상, 전력 소모 증가, 처리 지연이 발생하는 문제가 꾸준히 제기돼 왔다. 반면 인간의 망막은 빛을 감지하는 동시에 신경 신호를 ‘전처리’하는 고효율 구조를 갖는다.
연구팀은 이러한 망막 메커니즘에서 착안해, 태양전지가 빛을 전기 신호로 변환하고(1단), 해당 전기 신호가 시냅스 트랜지스터의 입력으로 다시 사용되는(2단) 캐스케이드 시냅스 메커니즘을 제안했다. 특히 태양전지-전기이중층(EDL) 커패시터 결합 구조에서 생성되는 전기 신호 자체가 PPF(짧은 시간 간격 자극이 누적돼 반응이 커지는 현상) 특성을 자연스럽게 포함해, 신호 전처리 단계에서부터 생물학적 시냅스와 유사한 시간적 누적 효과를 구현했다.
그 결과 기존 단일 시냅스 소자 대비 신호 증폭률과 기억 특성을 크게 향상시키는 동시에, 선형적인 가중치 변화를 통해 뉴로모픽 학습의 정확도 또한 개선했다. 나아가 키리가미 구조로 구현한 곡면 설계를 통해 인간 눈과 유사한 형태의 뉴로모픽 이미징 시스템을 실현했다.

【캐스케이드 뉴로모픽 구조의 개념 및 공정 구현】
실리콘 태양전지 기반 광수용부–산화물 반도체 시냅스 트랜지스터 직렬 캐스케이드(광-전자-시냅스) 뉴로모픽 구조의 개념과 공정 흐름도를 보여주는 그림.
(상단 좌측) 생물학적 시각 경로의 광-전기 변환 및 시냅스 전달·처리 과정을 개념적으로 도식화하고,
이를 모사해 실리콘 태양전지를 인공 광수용기로 사용해 생성 신호를 하부 시냅스 트랜지스터 입력으로 전달하는 구조를 제안함.
(상단 우측) 태양전지의 광유도 전압이 이미 시간적·전하적 전처리된 형태로 시냅스 트랜지스터 게이트에 인가되어,
추가 증폭 없이 PSP와 PSC를 효과적으로 유도하는 캐스케이드 원리를 회로 개념도로 설명한 그림.
(하단) 제안된 캐스케이드 구조를 표준 반도체 공정으로 구현한 결과를 제시함.
실리콘 태양전지-절연층-In₂O₃채널-금속 전극 적층 구조의 분해도와 소자 광학현미경 이미지, A-A′단면도를 통해
층 두께·정렬·전기적 연결을 확인함. 단일 플랫폼에서 광감지-신호 증폭-시냅스 동작을 통합해 뉴로모픽 이미징/인지 소자로의 확장 가능성을 보여줌.
한국을 비롯해 미국·중국·홍콩 등 글로벌 연구진이 참여한 이번 연구 성과는 향후 차세대 인공지능 카메라, 자율주행 및 로봇 비전, 인간-기계 인터페이스(HMI), 인공 시각 보조(시각 보철) 기술 등 다양한 분야에서 저전력·고효율 뉴로모픽 비전 플랫폼으로 활용될 것으로 기대된다.
심현석 교수는 “이번 연구는 소자 구조와 신호 전달 방식을 통합적으로 설계한 뉴로모픽 비전 플랫폼을 제안한 것으로, 인간의 시각 경로(망막-뇌)처럼 ‘보고, 처리하고, 저장하는’ 전자 시스템 구현에 한 걸음 더 다가갈 수 있게 됐다”고 말했다.
이번 논문은 전기전자공학부 심현석 교수가 제1저자, 미국 일리노이대 어바나-샴페인(University of Illinois Urbana-Champaign, UIUC) 위춘장(Cunjiang Yu) 교수가 교신저자로 수행했으며, 세계적인 학술지 『네이처』의 자매지인 『Nature Electronics』 온라인판 1월 7일자에 게재됐다.
- 논문 제목: A neuromorphic imager based on a cascaded optoelectronic synapse(캐스케이드 구조의 광전자 시냅스를 이용한 뉴로모픽 이미지 센서)
- 논문 링크: https://doi.org/10.1038/s41928-025-01540-w
※ 용어 설명
- 뉴로모픽(Neuromorphic): 뇌/신경계의 정보 처리 방식을 본떠, 적은 전력으로 빠르게 계산·학습하려는 기술/소자 설계 개념
- 시냅스 촉진(Synaptic facilitation): 짧은 시간 간격으로 자극이 반복되면 신호 전달이 더 강해지는 현상(‘누적 효과’)
- PPF(Paired-Pulse Facilitation): 두 번의 자극이 가까운 시간 간격으로 들어올 때 두 번째 반응이 더 커지는 시냅스 특성
- 전기이중층(EDL, Electric Double Layer): 전해질-전극 경계에 전하가 얇게 쌓이며 큰 정전용량을 만드는 구조(작은 전압으로도 신호 변화를 크게 만들 수 있음)
- 캐스케이드(cascaded) 구조: 2단 이상 기능을 직렬로 연결해, 앞단 출력이 뒷단 입력으로 이어지는 구성(여기서는 ‘빛→전기→시냅스’ 연쇄)
- 키리가미(kirigami) 구조: 종이를 자르고 펼치듯, 절개 패턴을 이용해 평면 재료를 잘 휘어지게 만들어 곡면 형태로 구현하는 설계 방식
[Abstract]
The human retina can provide inspiration for the development of optoelectronic devices for artificial vision systems. It has, in particular, a curved geometry for low optical aberration imaging and is capable of signal preprocessing, in part due to its high synaptic facilitation. However, it remains challenging to create artificial synaptic devices with mechanical softness, curvilinear form factors and superior synaptic facilitation. Here we report a cascaded two-stage optoelectronic synapse that uses silicon photovoltaic cells to modulate sodium-alginate-gated synaptic transistors. The photovoltaic cells and an electric double-layer capacitor convert the initial light signal into a gate voltage (stage one), which then controls the postsynaptic current in the channel of the synaptic transistor (stage two). This cascaded synaptic signal transmission mechanism results in high synaptic facilitation of the postsynaptic signal. The system exhibits stable and long-term linearly potentiated characteristics, enhancing the accuracy of pattern recognition. We use an array of the cascaded optoelectronic synapses to create a curvy, kirigami-structured neuromorphic imager, which mimics the curved geometry and neural signal transmission of the human retina and provides visual information sensing and preprocessing functions.
- Author (Pusan National University): Hyunseok Shim (School of Electrical and Electronics Engineering)
- Title of original paper: A neuromorphic imager based on a cascaded optoelectronic synapse
- Journal: Nature Electronics
- Web link: https://www.nature.com/articles/s41928-025-01540-w
- Contact e-mail: hshim@pusan.ac.kr
-
빛을 전기 신호로 증폭·기억하는 인공 시각 소자 개발
전기전자공학부/_UPLOAD/IMAGE/Board/68/2026/3/vv4pGIWfDN584ggj.JPG전기전자공학부심현석 교수팀, 사람처럼 보고 처리하고 기억하는 이미지 센서 구현빛을 전기 신호로 증폭·기억하는 인공 시각 소자 개발
부산대 국제공동연구팀이 사람 눈의 망막처럼 빛을 보자마자 신호를 스스로 키우고 기억하는 새로운 이미지 센서를 개발했다. 인간 망막의 신경 신호 전달 방식을 모사해 빛을 감지함과 동시에 신호를 증폭하고 기억하는 방식으로, 향후 인공지능 카메라와 자율주행·로봇 비전, 인공 시각 보조 기술 등 다양한 활용이 기대된다.전기전자공학부 심현석(사진) 교수 국제공동연구팀은 인간의 시각 경로(망막-뇌)처럼 빛을 ‘보고-처리하고-기억하는’ 기능을 하나의 구조에서 동시에 수행하는 차세대 곡면형 뉴로모픽(Neuromorphic·뇌신경모사형) 이미징 소자를 개발했다. 이번 성과는 기존 카메라 센서가 촬영(감지)과 처리·기억이 분리돼 전력 소모와 데이터 병목이 컸던 한계를 극복하고, 미래 저전력·고효율 인공지능 비전 시스템 구현을 앞당길 핵심 기술로 주목된다.
연구팀은 실리콘 태양전지(silicon solar cell)와 전해질 기반 시냅스 트랜지스터를 직렬로 연결한 캐스케이드(cascade) 광-전(optical-electrical) 시냅스 구조를 통해, 인간 망막에서 나타나는 시냅스 촉진(synaptic facilitation) 특성을 높은 수준으로 구현했다. 이 구조를 다수 집적하고, 키리가미(kirigami, 절개·접기) 구조 기반 설계를 적용해 사람 눈과 유사한 곡률의 곡면 뉴로모픽 이미저를 제작함으로써, 기존 이미지 센서와 달리 빛 감지와 동시에 신호 전처리 및 기억 기능 수행이 가능함을 실험으로 입증했다.
일반적인 이미지 센서는 빛을 전기 신호로 변환하는 역할에 주로 머물며, 인식·판단 등 고차 처리는 별도의 인공지능 프로세서가 담당한다. 이 과정에서 데이터 이동이 과도해져 병목 현상, 전력 소모 증가, 처리 지연이 발생하는 문제가 꾸준히 제기돼 왔다. 반면 인간의 망막은 빛을 감지하는 동시에 신경 신호를 ‘전처리’하는 고효율 구조를 갖는다.
연구팀은 이러한 망막 메커니즘에서 착안해, 태양전지가 빛을 전기 신호로 변환하고(1단), 해당 전기 신호가 시냅스 트랜지스터의 입력으로 다시 사용되는(2단) 캐스케이드 시냅스 메커니즘을 제안했다. 특히 태양전지-전기이중층(EDL) 커패시터 결합 구조에서 생성되는 전기 신호 자체가 PPF(짧은 시간 간격 자극이 누적돼 반응이 커지는 현상) 특성을 자연스럽게 포함해, 신호 전처리 단계에서부터 생물학적 시냅스와 유사한 시간적 누적 효과를 구현했다.
그 결과 기존 단일 시냅스 소자 대비 신호 증폭률과 기억 특성을 크게 향상시키는 동시에, 선형적인 가중치 변화를 통해 뉴로모픽 학습의 정확도 또한 개선했다. 나아가 키리가미 구조로 구현한 곡면 설계를 통해 인간 눈과 유사한 형태의 뉴로모픽 이미징 시스템을 실현했다.

【캐스케이드 뉴로모픽 구조의 개념 및 공정 구현】실리콘 태양전지 기반 광수용부–산화물 반도체 시냅스 트랜지스터 직렬 캐스케이드(광-전자-시냅스) 뉴로모픽 구조의 개념과 공정 흐름도를 보여주는 그림.
(상단 좌측) 생물학적 시각 경로의 광-전기 변환 및 시냅스 전달·처리 과정을 개념적으로 도식화하고,
이를 모사해 실리콘 태양전지를 인공 광수용기로 사용해 생성 신호를 하부 시냅스 트랜지스터 입력으로 전달하는 구조를 제안함.(상단 우측) 태양전지의 광유도 전압이 이미 시간적·전하적 전처리된 형태로 시냅스 트랜지스터 게이트에 인가되어,
추가 증폭 없이 PSP와 PSC를 효과적으로 유도하는 캐스케이드 원리를 회로 개념도로 설명한 그림.(하단) 제안된 캐스케이드 구조를 표준 반도체 공정으로 구현한 결과를 제시함.
실리콘 태양전지-절연층-In₂O₃채널-금속 전극 적층 구조의 분해도와 소자 광학현미경 이미지, A-A′단면도를 통해
층 두께·정렬·전기적 연결을 확인함. 단일 플랫폼에서 광감지-신호 증폭-시냅스 동작을 통합해 뉴로모픽 이미징/인지 소자로의 확장 가능성을 보여줌.한국을 비롯해 미국·중국·홍콩 등 글로벌 연구진이 참여한 이번 연구 성과는 향후 차세대 인공지능 카메라, 자율주행 및 로봇 비전, 인간-기계 인터페이스(HMI), 인공 시각 보조(시각 보철) 기술 등 다양한 분야에서 저전력·고효율 뉴로모픽 비전 플랫폼으로 활용될 것으로 기대된다.
심현석 교수는 “이번 연구는 소자 구조와 신호 전달 방식을 통합적으로 설계한 뉴로모픽 비전 플랫폼을 제안한 것으로, 인간의 시각 경로(망막-뇌)처럼 ‘보고, 처리하고, 저장하는’ 전자 시스템 구현에 한 걸음 더 다가갈 수 있게 됐다”고 말했다.
이번 논문은 전기전자공학부 심현석 교수가 제1저자, 미국 일리노이대 어바나-샴페인(University of Illinois Urbana-Champaign, UIUC) 위춘장(Cunjiang Yu) 교수가 교신저자로 수행했으며, 세계적인 학술지 『네이처』의 자매지인 『Nature Electronics』 온라인판 1월 7일자에 게재됐다.
- 논문 제목: A neuromorphic imager based on a cascaded optoelectronic synapse(캐스케이드 구조의 광전자 시냅스를 이용한 뉴로모픽 이미지 센서)
- 논문 링크: https://doi.org/10.1038/s41928-025-01540-w
※ 용어 설명
- 뉴로모픽(Neuromorphic): 뇌/신경계의 정보 처리 방식을 본떠, 적은 전력으로 빠르게 계산·학습하려는 기술/소자 설계 개념
- 시냅스 촉진(Synaptic facilitation): 짧은 시간 간격으로 자극이 반복되면 신호 전달이 더 강해지는 현상(‘누적 효과’)
- PPF(Paired-Pulse Facilitation): 두 번의 자극이 가까운 시간 간격으로 들어올 때 두 번째 반응이 더 커지는 시냅스 특성
- 전기이중층(EDL, Electric Double Layer): 전해질-전극 경계에 전하가 얇게 쌓이며 큰 정전용량을 만드는 구조(작은 전압으로도 신호 변화를 크게 만들 수 있음)
- 캐스케이드(cascaded) 구조: 2단 이상 기능을 직렬로 연결해, 앞단 출력이 뒷단 입력으로 이어지는 구성(여기서는 ‘빛→전기→시냅스’ 연쇄)
- 키리가미(kirigami) 구조: 종이를 자르고 펼치듯, 절개 패턴을 이용해 평면 재료를 잘 휘어지게 만들어 곡면 형태로 구현하는 설계 방식
[Abstract]
The human retina can provide inspiration for the development of optoelectronic devices for artificial vision systems. It has, in particular, a curved geometry for low optical aberration imaging and is capable of signal preprocessing, in part due to its high synaptic facilitation. However, it remains challenging to create artificial synaptic devices with mechanical softness, curvilinear form factors and superior synaptic facilitation. Here we report a cascaded two-stage optoelectronic synapse that uses silicon photovoltaic cells to modulate sodium-alginate-gated synaptic transistors. The photovoltaic cells and an electric double-layer capacitor convert the initial light signal into a gate voltage (stage one), which then controls the postsynaptic current in the channel of the synaptic transistor (stage two). This cascaded synaptic signal transmission mechanism results in high synaptic facilitation of the postsynaptic signal. The system exhibits stable and long-term linearly potentiated characteristics, enhancing the accuracy of pattern recognition. We use an array of the cascaded optoelectronic synapses to create a curvy, kirigami-structured neuromorphic imager, which mimics the curved geometry and neural signal transmission of the human retina and provides visual information sensing and preprocessing functions.
- Author (Pusan National University): Hyunseok Shim (School of Electrical and Electronics Engineering)
- Title of original paper: A neuromorphic imager based on a cascaded optoelectronic synapse
- Journal: Nature Electronics
- Web link: https://www.nature.com/articles/s41928-025-01540-w
- Contact e-mail: hshim@pusan.ac.kr
-
주택 소유의 장·단기 효과, 소득·생애주기별 실증 분석
도시공학과/_UPLOAD/IMAGE/Board/68/2026/2/4fm6xYexmSeXkQJI.JPG도시공학과최열 명예교수팀 "집을 사면 더 행복해질까"주택 소유의 장·단기 효과, 소득·생애주기별 실증 분석
집을 사면 더 행복해질까? 도시공학과 최열 명예교수 연구팀의 분석 결과, 주택 소유가 삶의 만족도에 미치는 영향은 보편적이지 않으며 소득 수준과 생애주기에 따라 상이한 양상을 보이는 것으로 나타났다.특히 저소득 가구의 경우 장기적인 자가 거주는 삶의 만족도를 높이는 반면, 임차에서 자가로 전환되는 단기 시점에서는 재정 부담과 스트레스로 오히려 만족도가 낮아지는 것으로 분석됐고, 중·고소득 가구에서는 장기 자가 거주가 삶의 만족도와 부정적인 관계를 보였다.
연구팀은 2008년부터 2023년까지 16년에 걸친 한국복지패널(KOWEPS) 장기 추적자료를 활용해 주택 소유의 단기 효과와 장기 효과를 구분해 분석한 논문을 발표했다.
주택은 단순한 거주 공간을 넘어 개인의 안정성, 자산 형성, 삶의 질을 좌우하는 핵심 요소로 인식돼 왔다. 특히 우리 사회에서는 ‘내 집 마련’이 삶의 만족과 직결되는 것으로 여겨지며, 주거정책 역시 자가 보유 확대를 주요 목표로 설정해 왔다. 그러나 주택 소유가 실제로 모든 계층과 생애주기에서 동일한 삶의 만족으로 이어지는지에 대해서는 장기적·실증적 검토가 충분히 이뤄지지 않았다.
기존 연구들은 주택 소유 여부와 삶의 만족도의 관계를 단면자료에 기반해 분석하거나, 자가와 임차를 단순 비교하는 데 그쳐 주택 소유의 시간적 효과와 집단 간 이질성을 충분히 설명하지 못했다. 주택 구입 과정에서 발생하는 재정적 부담과 스트레스가 단기적으로 삶의 만족도에 어떤 영향을 미치는지, 그리고 장기적인 주거 안정이 실제로 누구에게 긍정적으로 작용하는지에 대한 실증 연구는 매우 제한적이었다.
이에 연구팀은 동일 개인을 장기간 추적할 수 있는 패널자료의 강점을 활용해, 주택 소유의 ‘단기 전환 효과’와 ‘장기 지속 효과’를 명확히 구분하는 분석틀을 적용했다. 연구팀은 최소 10년 이상 조사에 참여한 2,860명의 개인을 대상으로, 순서형 패널 로짓 모형과 Mundlak 보정을 활용해 개인 내 변화와 개인 간 구조적 차이를 동시에 고려하는 분석을 수행했다.
연구 결과, 전체 표본에서는 주택 소유 여부 자체가 삶의 만족도와 통계적으로 유의한 관련을 보이지 않았다. 그러나 소득 수준별 분석에서는 뚜렷한 차이가 나타났다.
저소득 가구의 경우, 장기간 자가 상태를 유지하는 것은 삶의 만족도를 높이는 반면, 자가로 전환되는 단기 시점에서는 만족도가 유의하게 낮아졌다. 이는 주택 구입 과정에서 수반되는 대출 부담과 재정적 압박이 단기적으로 작용한 결과로 해석된다.
반면 중·고소득 가구에서는 장기적인 자가 거주 상태 자체가 삶의 만족도와 오히려 부정적인 관계를 보이며, 주택 소유가 반드시 주관적 행복으로 이어지지 않는 양상이 확인됐다. 이는 시간이 지남에 따라 주거 수준에 대한 기대치가 높아지는 현상과 관련된 결과로 해석된다.

【소득 수준별 주택 소유의 단기·장기 효과 방향】
주택 소유가 삶의 만족도에 미치는 영향은 소득 수준에 따라 상이하게 나타남.
저소득 가구의 경우 자가 전환 직후에는 만족도가 감소하지만, 장기적으로는 자가 상태가 삶의 만족도를 유의미하게 높이는 반면,
중·고소득 가구에서는 장기 자가 상태가 오히려 삶의 만족도와 부정적인 관계를 보임.한편, 이번 연구에서 가장 일관되게 강한 영향을 미친 요인은 주택 소유 여부가 아니라 근린 만족도였다. 거주 지역의 환경과 생활 여건에 대한 만족도가 높을수록 삶의 만족도 역시 크게 높아졌으며, 이러한 효과는 소득 수준과 생애주기에 관계없이 전 집단에서 공통적으로 관찰됐다. 이는 주거정책이 소유 중심 접근을 넘어, 지역 환경과 생활 여건의 질을 함께 고려해야 함을 시사한다.

【삶의 만족도 주요 결정 요인의 단기·장기 효과 비교】근린 만족도는 단기와 장기 모두에서 삶의 만족도와 가장 강한 양(+)의 관계를 보이는 반면,
주택 소유 여부 자체는 유의미한 영향을 미치지 않음.
장기적으로는 과도한 주거비 부담이 삶의 만족도를 유의하게 저하시키는 핵심 요인으로 확인됨.
이에 대해 최열(교신저자) 교수와 손희주(제1저자) 박사는 “이번 연구는 주택 소유의 효과가 획일적이지 않다는 점을 장기간 실증자료를 통해 확인했다는 점에서 의미가 크다”며 “주거정책은 단순히 자가 보유를 장려하는 방향이 아니라, 소득 수준과 생애주기에 따라 주거 안정이 실제 삶의 만족으로 이어질 수 있는 조건을 정교하게 설계할 필요가 있다”고 설명했다.
이번 연구에서 제시한 분석 방법과 결과는 주택 소유의 사회적 효과를 보다 입체적으로 이해할 수 있는 실증적 근거를 제공하며, 향후 주거복지 정책과 주택 공급 전략을 수립하는 데 있어 중요한 참고자료로 활용될 수 있을 것으로 기대된다.
이번 연구는 도시·주거 분야의 대표적인 국제 SSCI 학술지인 『도시과학국제저널(International Journal of Urban Sciences)』 2026년 6월호에 게재될 예정이다.
- 논문 제목: The homeownership effect: understanding short- and long-term impacts on life satisfaction(주택 소유 효과: 삶의 만족도에 대한 단기 및 장기적 영향)
- 논문 링크(DOI): https://doi.org/10.1080/12265934.2025.2570435
* 상단 연구진 사진: 왼쪽부터 최열 명예교수, 손희주 박사.
[Abstract]
While housing tenure has long been considered a key determinant of life satisfaction, its effects remain inconsistent across socioeconomic and demographic groups. Drawing on longitudinal data from the Korea Welfare Panel Study (KOWEPS), this study applies an ordered logistic panel model to examine how the relationship between housing tenure and life satisfaction varies by income level and life stage. Contrary to the common assumption that homeownership universally enhances well-being, the analysis finds no significant tenure effect in the full sample.
However, disaggregated results reveal important heterogeneity. Among low-income households, long-term homeownership is positively associated with life satisfaction, while short-term transitions are linked to reduced well-being – likely due to debtrelated stress. In contrast, tenure has negligible or even negative effects among middle – and high-income groups. A life-course perspective further indicates that tenure benefits are most salient in midlife, with limited effects in early or later adulthood.
Neighborhood satisfaction consistently emerges as a stronger predictor of well-being than tenure status itself, especially for low-income individuals. Moreover, a counterintuitive negative association is found between long-term housing adequacy and life satisfaction, particularly among higher-income respondents, reflecting rising expectations over time. These findings highlight the conditional and dynamic nature of housing effects on wellbeing.
Rather than promoting homeownership as a universal ideal, housing policy should be tailored to reflect the diverse needs of different socioeconomic and life-stage groups. The study contributes to the literature by integrating theoretical insights with panel-based empirical analysis, offering practical implications for more equitable and context-sensitive housing strategies.
- Author (Pusan National University): Yeol Choi (Department of Urban Planning and Engineering)
- Title of original paper: The homeownership effect: understanding short- and longterm impacts on life satisfaction
- Journal: International Journal of Urban Sciences
- Web link: https://www.tandfonline.com/doi/full/10.1080/12265934.2025.2570435
- Contact e-mail: yeolchoi@pusan.ac.kr
-
양자점 손상 최소화로 AR/VR용 초고해상도·고신뢰성 마이크로디스플레이 구현 청...
전기전자공학부/화학과/_UPLOAD/IMAGE/Board/68/2026/2/mwMaYSWuTuZN0tEx.JPG전기전자공학부/화학과노정균·황도훈 교수팀, 포토레지스트·리간드 교환 없이 양자점 초정밀 패터닝 성공양자점 손상 최소화로 AR/VR용 초고해상도·고신뢰성 마이크로디스플레이 구현 청신호부산대 연구팀이 차세대 디스플레이 소재인 양자점을 손상 없이 초정밀하게 배열할 수 있는 새로운 제조기술을 개발해, AR/VR용 초고해상도·고신뢰성 마이크로디스플레이 구현에 중요한 돌파구를 마련했다.
전기전자공학부 노정균 교수와 화학과 황도훈 교수 연구팀은 성균관대와의 공동연구를 통해 포토레지스트(photoresist, PR)와 리간드 교환 과정 없이 양자점(Quantum Dot, QD)을 정밀하게 미세 패터닝할 수 있는 새로운 비파괴형 포토리소그래피 기술을 개발했다.
양자점 발광다이오드(QD-LED)는 높은 색 순도와 용액공정 호환성으로 차세대 디스플레이 소자로 주목받고 있으나, RGB 픽셀을 고해상도로 구현할 수 있는 패터닝 기술 부족이 상용화의 가장 큰 걸림돌이었다. 잉크젯 프린팅 공정은 초고해상도 픽셀 형성에 적합하지 않으며, 포토리소그래피 기반 패터닝 방식은 공정 중 양자점 손상이 많이 발생하거나 복잡한 리간드 교환 공정을 필요로 하는 등 한계를 가지고 있다.
반도체나 디스플레이를 만들 때는 아주 작은 패턴을 정확하게 새기는 기술이 필수적이다. 이를 위해 보통 ‘포토레지스트(PR)’라는 감광재(感光材)를 도포한 뒤 빛을 쏘여 원하는 부분만 남기는 포토리소그래피 공정을 거친다. 그러나 PR을 바르고 제거하는 과정이 복잡하고 비용이 크며, 양자점처럼 민감한 소재에는 손상을 줄 위험이 크다는 문제가 있다.
양자점의 경우에는 PR을 직접 사용하는 대신, 양자점 표면 리간드를 교환해 포토리소그래피에 활용하는 'PR-프리' 공정들이 보고되기도 했다. 그러나 이러한 방식은 복잡한 리간드 교환 공정을 도입해야 하고, 이 과정에서 양자점 표면 결함 증가 등으로 인해 원래의 우수한 광학 특성이 저하되는 문제가 있다.
이에, 연구팀은 PR과 리간드 교환 과정 없이 양자점의 특성을 해치지 않고(비파괴) 정밀하게 배열하는 기술을 개발했다. 공정은 단순화하면서 소재 손상을 최소화해 차세대 디스플레이 핵심소자인 양자점 발광다이오드(QD-LED)의 초고해상도 구현과 함께 효율·수명 향상을 동시에 이룬 공정 혁신으로 평가된다.
연구팀이 개발한 것은 양자점을 한 자리에 고정시키면서도 손상 없이 미세하게 배열할 수 있는 ‘블렌드형 발광층(blended Emissive Layer, b-EML) 기반 고해상도 패터닝 기술’이다.

【양자점-광가교형 고분자 블렌드형 발광층을 이용한 양자점 고해상도 패터닝 기법】
이 기술은 양자점을 정공수송층 고분자(PVK)와 광가교제(FPA)와 섞은 뒤 자외선을 쏘이면, 고분자끼리 서로 단단히 연결돼 3차원 그물망 구조를 만들고, 그 사이에 양자점이 안정적으로 고정되는 원리다. 이 과정을 통해 원하는 위치에만 양자점을 정확하게 남길 수 있어 매우 높은 해상도의 미세 패턴 구현이 가능하다.
특히, 양자점 자체를 경화시키는 방식이 아니라 주변 고분자만 경화시키는 방식으로, 첨가제를 최소화하면서도 양자점 손상을 효과적으로 막을 수 있다는 장점이 있다.
이 기술을 통해 연구팀은 단색 10,000 ppi, RGB 풀컬러 1,000 ppi 이상의 초고해상도 양자점 패턴을 구현했으며, 포토레지스트·리간드 교환 없이도 높은 패턴 충실도와 발광 특성 보존성을 동시에 확보했다.
또한 개발된 패터닝 방식을 적용해 양자점 발광다이오드를 제작한 결과, 블렌드형 발광층을 통한 전자 과주입 억제와 정공주입 향상의 효과로 외부양자효율이 1.7배 향상되고 구동 수명이 3배 증가함을 확인했다.

【차세대 디스플레이용 광가교 양자점(QD) 패턴】
이번 연구는 QD 패터닝과 소자 안정성 간의 상충관계를 해소한 보편적·저손상형 QD 포토리소그래피 기술로, 공정 단순화와 성능 향상을 동시에 달성했다는 점에서 큰 의미를 갖는다.
또한 해당 패터닝 기술은 유해 중금속인 카드뮴을 쓰지 않는 InP(인듐 포스파이드) 기반 탈카드뮴 양자점, 은 나노입자 등 다양한 나노결정 재료에도 적용 가능함을 실험적으로 확인해, 차세대 디스플레이 및 광전자 소자 분야에서 폭넓은 응용 가능성을 보여준다.
연구를 주도한 노정균 교수는 “이번에 개발된 양자점 패터닝 기술은 공정 중 양자점의 손상을 방지하면서도 간단하고 다양한 양자점 시스템에 적용 가능한 높은 범용성을 가진다는 점이 특징”이라며 “이 외에도 양자점 발광다이오드의 수명 및 효율 향상과 공정 단순화를 가능하게 한다는 점에서 양자점 기반의 AR/VR용 디스플레이 상용화를 앞당길 수 있을 것”이라고 밝혔다.

【저널 표지에 소개된 연구팀의 논문 이미지】
이번 연구는 전기전자공학부 이재엽 박사과정생과 화학과 곽선이 박사과정생이 공동 제1저자, 부산대 노정균 교수와 황도훈 교수, 성균관대 임재훈 교수가 공동 교신저자로 수행했다.
해당 연구결과는 재료과학 분야의 국제 저명 학술지 『어드밴스드 펑셔널 머티리얼즈(Advanced Functional Materials)』 2월호에 게재됐으며, 연구의 창의성과 실용적 완성도를 인정받아 표지논문(Inside front cover)으로 선정됐다.
- 논문 제목: Universal and Nondestructive Direct Photolithography of Colloidal Quantum Dots Using Photocrosslinkable Polymer Blends(광가교성 고분자 블렌드를 이용한 콜로이드 양자점의 범용적이고 비파괴적인 직접 포토리소그래피)
- 논문 링크: https://doi.org/10.1002/adfm.202520047
해당 연구는 한국연구재단의 글로컬R&D 지원사업, 중견연구자 지원사업, 박사과정생연구장려금 지원사업, 국가전략기술소재개발 사업, 한국산업기술기획평가원의 디스플레이혁신공정플랫폼구축 사업, 부산광역시·교육부 RISE 사업의 지원으로 수행됐다.
* 상단 연구진 사진: 왼쪽부터 노정균 교수, 황도훈 교수.
[Abstract]
Researchers Uncover Scalable Method for Ultrahigh-Resolution Quantum Dot Displays
Colloidal quantum dot (QD) light-emitting diodes have great potential in display applications. However, their commercialization remains a challenge due to the difficulty in achieving high-resolution patterning of QDs without degrading their optical properties. To address this, researchers have developed a nondestructive method for ultrahigh-resolution QD patterning. By blending QDs with a photocrosslinkable polymer, the approach preserves their optical properties and boosts efficiency and lifetime, paving the way for development of next-generation display technologies.
Over the past decade, colloidal quantum dots (QDs) have emerged as promising materials for next-generation displays due to their tunable emission, high brightness, and compatibility with low-cost solution processing. However, a major challenge is, achieving ultrahigh- resolution patterning without damaging their fragile surface chemistry. Existing methods such as inkjet printing and photolithography-based processes either fall short in resolution or compromise QD performance.
To address this, a research team led by Associate Professor Jeongkyun Roh from the Department of Electrical Engineering, Pusan National University, Republic of Korea, has introduced a universal, photoresist-free, and nondestructive direct photolithography method for QD patterning. Instead of exposing QDs to harsh chemical modifications, the team engineered a photocrosslinkable blended emissive layer (b-EML). This layer is formed by mixing QDs with a hole-transport polymer and a small fraction of an ultraviolet (UV)-activated crosslinker, enabling precise patterning while preserving QD integrity. The study was published in the journal of Advanced Functional Materials on 29 September 2025.
The method works by blending QDs with photocrosslinkable hole-transport layer polymers, then exposing the film to UV light, which triggers crosslinking leading to a robust polymer network that immobilizes and protects the QDs. The patterned regions can then be developed simply using a solvent, producing high-resolution QD patterns.
“We have developed a simple blending approach that yields ultrahigh-resolution QD patterns compatible with demanding display applications. This nondestructive approach maintains QD performance, and provides resolutions exceeding 10,000 pixels per inch (ppi), as well as red, green, and blue full-color pixels above 1,000 ppi on a 4-inch wafer—well within the range required for emerging displays and other near-eye microdisplays,” says Dr. Roh.
The study demonstrated that this b-EML enables ultrahigh-resolution QD patterning while maintaining optical quality, improving charge balance, enhancing external quantum efficiency by 1.7-fold, and nearly tripling operational lifetime. This ultrahigh-resolution patterning directly benefits near-eye microdisplays for augmented reality (AR), virtual reality (VR), and smart glasses, where pixel densities above 1,000 ppi are essential. Additionally, the ability to co-pattern QDs with other nanocrystals opens avenues for integrated optoelectronic chips and multifunctional displays.
Overall, this work presents a simple, robust, and universally compatible solution to a major hurdle in QD-light-emitting diodes (LEDs) displays. By relying on a photocrosslinkable matrix rather than ligand exchange or photoresists, the method is nondestructive, preserves QD brightness, and boosts device efficiency and works across multiple nanocrystal systems.
“Our research can bridge the gap between today’s laboratory-scale QD-LED prototypes and tomorrow’s commercial QD-based displays. Technically, our photocrosslinkable b-EML approaches tackles several long-standing bottlenecks for high-resolution QD patterning at once,” concludes Dr. Roh.
- Authors (Pusan National University)
· Jaeyeop Lee, Jeongkyun Roh (Department of electrical engineering)
· Seon Lee Kwak, Do-Hoon Hwang (Department of Chemistry and Chemistry Institute for Functional Materials)
- Title of original paper: Universal and Nondestructive Direct Photolithography ofColloidal Quantum Dots Using Photocrosslinkable PolymerBlends
- Journal: Advanced Functional Materials
- Web link: https://doi.org/10.1002/adfm.202520047
- Contact e-mail: jkroh@pusan.ac.kr
-
비열 효과 미세조직 가속 영향 규명
기계공학부/_UPLOAD/IMAGE/Board/68/2026/2/ckAxtPA546BkJpGr.JPG기계공학부이태경 교수팀, T-type 시편으로 전류펄스 열·비열 효과 분리 성공비열 효과 미세조직 가속 영향 규명기계공학부 이태경(사진) 교수 연구팀이 전류펄스 인가 처리(EPT)*에서 줄열(Joule heat, 열 효과)**과 전류 자체가 유발하는 비열(athermal) 효과***를 단일 시편 내에서 분리·관찰할 수 있는 ‘T-type 시편’**** 기반의 독창적인 방법론을 개발했다. 연구팀은 이를 마그네슘 합금에 적용해 비열 효과가 미세조직 변화 속도를 유의미하게 가속한다는 사실도 입증했다.
* EPT: 전류펄스 인가 처리(electropulsing treatment). 금속 소재에 펄스 형태의 전류를 가해 소재의 미세조직 및 성능을 제어하는 기술.
** 줄열(Joule heat, 열 효과): 재료(도체)에 전류가 흐를 때 전기저항 때문에 전기에너지가 열로 변환돼 발생하는 발열. 19세기 영국의 물리학자 제임스 프레스콧 줄(James Prescott Joule)이 전류에 의한 저항 발열의 관계(줄의 법칙)를 실험적으로 정립했으므로, 이 현상을 줄열(Joule heat)이라 부름.
*** 비열(athermal) 효과: 금속 소재에 전류가 흐를 때 발생하는 줄열(Joule heat)이 소재에 미치는 영향을 열 효과(thermal effect)라 하며, 그 외의 효과를 비열 효과(athermal effect)라 지칭.
**** T-type 시편: 현재까지의 EPT 연구는 I자 형상의 시편을 사용해 왔으나, 이번 연구에서는 T자 형상의 시편을 도입하고 EPT 공정 변수를 최적화해 시편의 특정 구간으로만 전류가 흐르도록 설계한 새로운 방법론을 개발.
‘전류펄스 인가 처리(EPT, Electropulsing Treatment)’는 금속 소재에 펄스(pulse, 순간 전류) 형태의 전류를 가해 열처리하는 기술로, 기존 노(爐) 열처리(furnace heat treatment)* 대비 에너지 효율이 높고 가열 속도가 빠르다는 점에서 차세대 금속 가공 기술로 주목받아 왔다. 그러나 EPT의 효과가 줄열에 의한 ‘열 효과’ 때문인지, 전류 자체가 만드는 ‘비열 효과’가 독립적으로 존재하는지에 대해서는 학계에서 오랜 논쟁이 이어져 왔다.
* 노(爐) 열처리(furnace heat treatment): 소재를 ‘가열로’에 넣어 일정 온도로 데운 뒤 잠시 유지하고 식히는 방식으로, 열만으로 재료 성질을 바꾸는 처리 방법.
이번 연구의 핵심은 기존의 I자형 시편 중심 실험에서 벗어나, 전류가 흐르는 경로와 열이 전달되는 경로를 구조적으로 분리할 수 있는 T자형(T-type) 시편을 설계했다는 데 있다. 연구팀은 EPT 공정 변수를 최적화해 시편의 특정 구간으로만 전류가 흐르도록 만들었고, 특정 조건에서는 같은 시편 내부에서 열 효과와 비열 효과를 분리해 비교할 수 있음을 확인했다.

【T-type 시편을 활용한 전류펄스의 열·비열 효과 분리 개념】
검증 실험은 마그네슘 합금 T-type 시편을 제작해 진행됐다. 시편 내 C지점에는 전류가 흐르도록, D지점에는 전류가 흐르지 않도록 설계한 뒤, 70~77A/mm² 수준의 전류펄스를 인가했을 때 두 지점이 동일 온도로 가열되는 조건을 구현했다. 즉, C와 D에서 열 효과의 크기를 동일하게 맞춘 것이다.
그럼에도 C지점에서는 D지점에 비해 미세조직 변화가 뚜렷하게 가속되는 현상이 확인됐다. 결정립 성장, 저경각계 소멸, 전위밀도 감소, 정적 재결정 등 다양한 미세조직 변화가 일관되게 빨라졌으며, 연구팀은 이를 C지점에만 독립적으로 작용한 비열 효과의 영향으로 해석했다. 전류로부터 추가적인 전기 에너지가 변형 유기 경계 이동(strain-induced boundary mechanism)에 구동력을 제공해 원자 확산과 결정립계 이동을 촉진했다는 설명이다.
이태경 교수는 “이번에 제안한 T-type 시편 방법론은 그동안 명확히 구분하기 어려웠던 EPT의 열·비열 효과를 단일 시편 내에서 분리해 내는 혁신적 시도”라며 “EPT 공정 메커니즘을 해석하는 표준 도구로 활용돼 금속 미세조직 제어 기술을 한 단계 도약시킬 수 있을 것으로 기대된다”고 말했다.
이번 연구는 기계공학부 이태경 교수가 교신저자, 송종한 연구원이 제1저자로 수행했으며, 부산대 기계공학부 백승훈 교수와 전기전자공학부 노정균 교수, 경북대 금속재료공학과 박성혁 교수가 공저자로 참여했다.
해당 연구는 국방기술진흥연구소(KRIT)와 한국에너지기술평가원(KETEP)의 지원을 받아 수행됐으며, 연구 결과는 국제학술지 『Journal of Magnesium and Alloys』 온라인판 2025년 12월 8일자에 게재됐다.
- 논문 제목: Validating the athermal contribution of electropulsing treatment utilizing T-type Mg specimen(T-type 시편 설계를 통한 전류펄스의 비열 효과 분리)
- 논문 링크: https://doi.org/10.1016/j.jma.2025.11.017
[Abstract]
Metal Design & Mechanics Lab (P.I.: Professor Taekyung Lee) successfully introduced a novel methodology, utilizing T-type specimen, to completely separate the thermal and athermal contributions of electropulsing treatment (EPT) within a single experiment. The key innovation was establishing two reference points (namely, points C and D) where temperatures remained comparable (<6 K deviation). Such a setup ensured that any difference in microstructural evolution arose solely from the athermal effects. The results consistently demonstrated that the region exposed to the electric current (i.e., point C) exhibited remarkably accelerated kinetics of microstructural evolution (e.g., a growth of preexisting grains, a reduction of low-angle grain boundaries, an annihilation of geometrically necessary dislcoations, and the extent of static recrystallization) compared to the heat-only counterpart (point D). This proved that the athermal EPT contribution provides an additional driving force to the strain-induced boundary migration mechanism, stimulating microstructural evolution beyond thermal effects alone. This T-type specimen offers a robust x-framework for decoupling EPT effects at the macroscale, providing a valuable tool for optimizing electrically-assisted forming and other high-efficiency manufacturing processes.
- Authors (Pusan National University): Taekyung Lee (corresponding) & Jong Han Song (1st) (School of Mechanical Engineering)
- Title of original paper: Validating the athermal contribution of electropulsing treatment utilizing T-type Mg specimen
- Journal: Journal of Magnesium and Alloys
- Web link: https://doi.org/10.1016/j.jma.2025.11.017
- Contact e-mail: taeklee@pnu.edu
-
'생각하는 하드웨어' 차세대 AI 엣지 비전 플랫폼 기대
물리학과/_UPLOAD/IMAGE/Board/68/2026/1/bA3Cj0IzHUwQOHt5.JPG물리학과김지희 교수팀, 전원 없이 빛으로 감지·연산하는 인센서 이미징 기술 구현'생각하는 하드웨어' 차세대 AI 엣지 비전 플랫폼 기대
물리학과 김지희 교수 연구팀이 전원 없이도 빛에 의해 스스로 반응하고 이미지를 처리하는 새로운 형태의 자가 구동형 인센서 이미징(in-sensor imaging) 기술을 구현했다.이번 연구는 빛의 세기와 방향뿐만 아니라 전류 응답의 부호를 제어해 감지와 연산이 하나의 소자에서 동시에 일어나는 ‘생각하는 하드웨어’의 가능성을 열었으며, 센서에서 직접 이미지 전처리를 수행할 수 있는 하드웨어적 기반을 구축함으로써 Vision(비전) AI 및 Edge(엣지) AI 기술과의 융합 가능성을 제시해 초저전력 인공지능(AI) 엣지 비전 기술로의 확장을 기대하게 한다.

【김지희 교수】
김지희 교수팀은 성균관대와의 공동연구로, 1차원 나노선 Nb2Pd3Se8과 2차원 나노면 WSe2를 결합한 새로운 반데르발스 이종접합 구조*를 이용해 게이트 전압**으로 전류 방향이 조절되는 자가 구동형 광검출기를 제작하고, 이를 인센서 이미지 처리*** 소자에 응용할 수 있음을 실험적으로 입증했다.
이번 연구에서 개발된 소자는 전원 없이도 빛에 의해 스스로 전류를 생성하며, 게이트 전압 조절을 통해 전류의 극성(양↔음)을 전환할 수 있는 것이 특징이다. 이러한 양·음 광전류 전환(polarity switching) 기능은 신호의 가중치를 조절하거나 이미지 필터링 연산에 활용할 수 있어, 기존 이미지 센서와 달리 감지와 계산을 하나의 장치에서 동시에 수행할 수 있는 기반을 마련했다.또한 연구팀은 1차원 나노선인 Nb2Pd3Se8의 좁은 밴드갭과 높은 비대칭성을 활용해 넓은 파장대(자외선~근적외선)에서 빠른 광응답을 구현했다.
실험 결과, 소자는 약 3마이크로초(백만분의 1초)의 초고속 응답 속도와 232 mA/W의 높은 광응답도(photoresponsivity)를 달성했으며, 외부 양자 효율 77%와 탐지율(Detectivity) 6.25×1010 Jones의 우수한 성능을 보였다.

【1D Nb2Pd3Se8/ 2D WSe2 이종접합 광검출기의 게이트 조절형 전류 방향 전환】
게이트 전압 변화에 따라 양·음 광전류가 전환되며, 인센서 이미지 처리에 활용 가능한 양방향 광응답을 보여줌.
이번 성과는 1차원 나노선과 2차원 나노면의 결합을 통해 차세대 반데르발스 이종접합 소자에서 광-전기 신호를 정밀하게 제어할 수 있음을 실험적으로 보여준 것으로, 나노전자 및 광컴퓨팅 분야에서 새로운 소자 설계 방향을 제시한다는 점에서 학문적 의미가 크다.
김지희 교수는 “이번 연구는 2차원 소재의 전자-광 특성을 정밀하게 제어해, 하나의 소자에서 감지와 연산이 동시에 이루어질 수 있음을 실험적으로 입증한 데 의의가 있다”며 “앞으로 이번 기술을 확장해 인공지능(AI) 하드웨어의 융합을 추진할 계획이다. 특히 인센서 이미지 처리 기술을 Vision AI와 Edge AI 시스템의 입력 단계에 적용해, 실시간 데이터 전처리·노이즈 제거·특징 추출을 수행하는 스마트 센서 플랫폼으로 발전시키려는 구상이다. 이는 자율주행, 로봇비전, 생체 모방 감각 시스템 등 미래 AI 융합산업의 핵심기술로 이어질 것으로 기대된다”고 설명했다.
해당 연구 결과는 재료과학 분야의 세계적 권위 학술지 『Advanced Materials』 2025년 10월 31일자에 게재됐다.
- 논문 제목: Van der Waals integration of 1D Nb2Pd3Se8 and 2D WSe2 for gate-tunable in-sensor imaging processing (1D-2D 반데르발스 이종접합을 이용한 게이트 조절형 인센서 이미지 처리 소자)
- 논문 링크: https://doi.org/10.1002/adma.202500011
※ 용어 설명
* 반데르발스 이종접합 구조: 원자층 물질을 서로 다른 종류로 적층해 약한 반데르발스 힘으로 결합시킨 구조로, 격자 불일치 없이 새로운 전자·광학적 특성을 구현할 수 있는 차세대 나노소자 구조임.
** 게이트 전압: 반도체 소자에서 채널의 전자 밀도를 바꿔주는 전압으로, 여기서 스위치이자 연산 조절기 역할을 함.
*** 인센서 이미지 처리: 빛을 감지하는 이미지 센서 내부에 감지와 연산이 동시에 이루어지는 기술로, 별도의 전력 공급이나 외부 프로세서 없이 초저전력 지능형 이미지 처리를 가능하게 함.
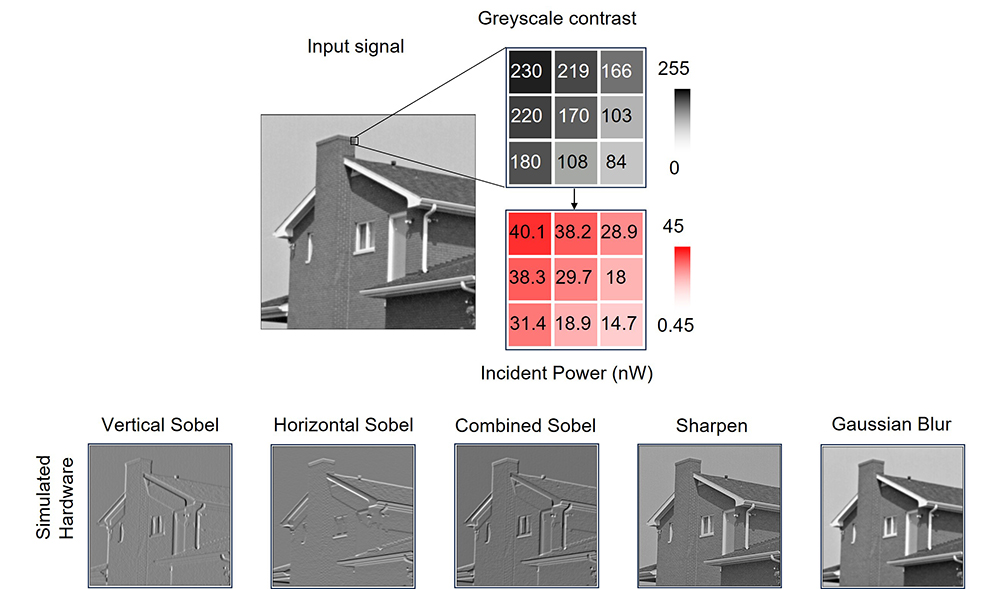
* 상단 연구 이미지: (위)레이저 세기를 아날로그 신호로 변환해 얻은 8비트 회색조 입력 이미지와 (아래)1D Nb2Pd3Se8/ 2D WSe2 이종접합 소자의 게이트 조절형 광응답(R값)을 이용한 인센서 이미지 처리 시뮬레이션 결과. 다양한 커널(Sobel, Sharpen, Gaussian Blur 등)을 적용해 엣지 검출 및 이미지 향상 기능을 구현함.
[Abstract]
1D and 2D integrations provide significant promise for machine vision by enabling compact, power-efficient optoelectronic devices. However, the potential of 1D materials in mixed-dimensional structures for convolutional image processing remains largely unexplored. Here, high-quality 1D- Nb2Pd3Se8 is synthesized and integrated with 2D-WSe2 to form self-powered photodetectors, exhibiting gate-tunable bi-directional photoresponse for image processing. Utilizing the narrow band gap and favorable work function of 1D- Nb2Pd3Se8, a type-I junction and 1D van der Waals interface are established with transition metal dichalcogenides. The gate tunable built-in electric field enables switching between n-p and n-n+ configurations, allowing the drift photocurrent direction to be reversed, achieving both negative and positive photocurrent. Furthermore, efficient conversion of high-energy photons along one dimension enhances sensitivity at 375 nm. The device achieves a responsivity of 232 mA W-1, external quantum efficiency of 77% at 375 nm illumination, rapid response time of ~3 µs, detectivity of 6.35 × 1010 Jones, and broadband photodetection from ultraviolet to near-infrared. The demonstrated gate-controllable, bi-directional photoresponse with linear power dependence in a 1D heterojunction offers a promising platform for in-sensor convolutional processing with high integration and portability.
- Author (Pusan National University): Ji-Hee Kim (Department of Physics)
- Title of original paper: Van der Waals integration of 1D Nb2Pd3Se8 and 2D WSe2 for gate-tunable in-sensor imaging processing
- Journal: Advanced Materials
- Web link: https://doi.org/10.1002/adma.202500011
- Contact e-mail: kimjihee@pusan.ac.kr
-
소형 전열가전 배출 초미세먼지 특성과 건강영향 평가 분석
환경공학과/_UPLOAD/IMAGE/Board/68/2026/1/2EY2jP1OOuPjkoqm.JPG환경공학과김창혁 교수팀 "생활 전열가전, 초미세먼지 배출…어린아이에게 더 유해할 수 있다"소형 전열가전 배출 초미세먼지 특성과 건강영향 평가 분석
흔히 사용하는 헤어드라이어와 에어프라이어, 토스터 등 생활 가전이 눈에 보이지 않는 ‘초미세먼지’를 내뿜고 있다면? 환경공학과 김창혁 교수 연구팀이 소형 전열가전제품에서 배출되는 초미세먼지 입자의 물리화학적 특성과 잠재적 건강 영향을 규명한 연구 결과를 발표해 주목받고 있다.해당 연구는 일상생활에서 흔히 사용하는 소형 전열가전제품에서 발생하는 직경 100nm 이하 초미세먼지 입자(UltraFine Particles, UFPs)의 물리화학적 특성을 분석하고, 폐침적 시뮬레이션 결과 기반 나이에 따른 UFP의 건강영향을 다뤘다.
연구팀은 국내 온라인 쇼핑 사이트에서 판매순위 최상위권을 차지하고 있는 소형 전열가전제품을 종류별로 선정해, 일상생활 중 제품을 사용하며 사용자가 제품에서 배출하는 UFP에 노출되는 환경을 모사하는 실험장치를 구성했다. 부피 1㎥의 LDPE 모사챔버를 제작하고, 챔버 중앙에서 제품에서 배출한 입자를 실시간으로 샘플링해 측정하면서, 동시에 입자를 포집해 물리화학적 특성을 분석했다.
※ 챔버(chamber): 연구실에서 실제 생활환경을 축소·재현하기 위해 만든 밀폐형 실험 공간.
기존의 초미세먼지 지표인 PM2.5(particulate matter, 직경 2.5 μm 이하 입자의 질량 농도)는 UFP와 같은 작은 입자의 건강영향을 과소평가할 수 있는 한계를 지니고 있다. 따라서, UFP 노출 지표로서 입자 수농도 크기분포(particle number size distributions, PNSDs)에 대해 U.S. EPA(U.S. Environmental Protection Agency, 미국 환경보호청)와 EEA(European Environment Agency, 유럽 환경청) 등에서 기준 측정시험법 제정 및 규제를 검토하는 등 국제사회의 관심이 높아지고 있다.
이에 연구팀은 SMPS(scanning mobility particle sizer)를 활용해 PNSDs를 실시간으로 측정하고, MPPD(multiple-path particle dosimetry) 입자 폐침적 시뮬레이션을 통해 연령대별 폐 모델에서 소형 전열가전제품 배출 입자의 노출 정도를 정량적 분석하고 비교했다.
관찰 결과, 헤어드라이어에서는 주로 직경 25nm 미만의 매우 작은 Nucleation mode 입자 배출이 지배적이었고, 에어프라이어와 토스터에서의 UFP는 상대적으로 큰 Accumulation mode 입자가 대다수 배출되는 경향을 보였다. Brushed DC motor와 Heating coil을 함께 사용하는 헤어드라이어는 Brushless DC motor(BLDC motor) 대비 배출입자 총수농도가 10~100배 높게 나타났으며, 에어프라이어와 토스터는 가전을 작동한 지 5분 안에 기하평균직경(Geometric mean diameter, GMD)이 빠르게 증가하고, 작동 온도를 높일수록 최종 총수농도 역시 큰 폭으로 증가했다.
TEM-EDS 분석에서는 헤어드라이어 사용 중 구리와 티타늄 등 중금속과 Tarball 형태의 탄소성 입자가 확인됐는데, 이는 Brushed moder 내부에서 Brush와 정류자 간의 마찰로 인한 마모와 스파크 발생이 중금속 입자를 배출하고, 더불어 실내공기 중 고분자 입자가 헤어드라이어 흡기로 유입, Heating coil 주변에서 가열돼 불완전 연소를 거치며 발생한 것으로 보인다. 니켈이 주성분인 Heating coil에서 배출하는 입자는 PNSD 측정 결과와 같이 25nm 이하로 만들어지기 쉬우므로, TEM-EDS로는 직접 분석하기 어렵지만 PNSD로 존재 가능성이 매우 높음을 알 수 있었다.

【소형 가전 사용 시 발생하는 초미세입자(UFP)의 생성·성분(금속/탄소계)과 폐 침착(어린이 > 성인) 특성】
MPPD 시뮬레이션에 따르면, 측정된 PNSD 범위의 UFP는 주로 폐포(Pulmonary) 영역에 대부분 침적되고, 기관지계(Trancheo-bronchial)에 약 4분의 1, 상기도(Head)에 나머지가 침적되는 경향을 보였다. 이 입자 폐침착 분포는 UFP가 폐와 기도를 중심으로 큰 영향을 미칠 수 있음을 의미하며, 일부는 후각 경로 등을 통해 중추신경계로의 전이도 가능할 수 있다는 점을 시사한다. 만약 폐세포 내에서 중금속이 용출돼 이온 형태로 전환될 경우 혈류를 통해 심장과 신장을 포함한 주요 신체 장기에 2차 영향을 줄 수 있으며, 성인 대비 폐의 체적이 적은 소아의 경우 성인보다 폐의 단위면적당 더 많은 입자 침적이 발생할 수 있어 건강영향이 급증할 수 있다.
이번 연구 결과는 실제 가정에서 널리 쓰이는 Brushed DC motor와 Heating coil 기반 소형 전열가전(헤어드라이어, 에어프라이어, 토스터)을 대상으로, 실시간 배출 입자 수농도 크기분포 측정과 오프라인 물리화학적 특성 분석, 그리고 MPPD 폐침적 시뮬레이션 결과를 종합적으로 판단해 UFP의 생성-성상-신체노출을 일관되게 추적하고 연령에 따른 입자 노출 특성 차이를 규명한 데 의의가 있다.
특히 헤어드라이어의 모터 유형(brushed DC vs BLDC)에 따라 배출 UFP의 총수농도와 수농도 크기분포의 변화가 발생함을 체계적으로 비교분석함으로써, 소비자의 선택과 제조사 설계 개선 등에 직접 활용 가능한 근거를 제시했다.
김창혁 교수는 “이번 연구는 실내 공기질의 질적 개선을 위해서는 외부 유입 차단뿐만 아니라 내부 오염원 관리가 병행돼야 하고, 건강 취약성을 고려한 연령대별 맞춤 기준이 요구된다는 점을 시사한다”고 말했다.
해당 연구는 환경·보건 분야의 국제 저명 학술지인 『Journal of Hazardous Materials』 2025년 10월 15일자(Vol. 498)에 게재됐다.
환경공학과 김창혁 교수(교신저자) 연구팀에서 류근우 석사과정생, 김정빈 석박통합과정생(사회환경시스템공학과 환경공학전공, 공동 제1저자)이 과학기술정보통신부의 재원으로 한국연구재단의 우수신진연구와 디지털 기반 기후변화 예측 및 피해 최소화 사업의 지원으로 연구를 수행했다.
- 논문 제목: Physicochemical characteristics and health impacts of ultrafine particles emitted from small home appliances equipped with heating coils and brushed motors(가열 코일과 브러시 모터를 장착한 소형 가전제품 배출 초미세 입자의 물리화학적 특성과 건강영향)
- 논문 링크: https://doi.org/10.1016/j.jhazmat.2025.139796
* 상단 연구진 사진: 왼쪽부터 류근우 석사과정생, 김정빈 석박통합과정생, 김창혁 교수.
[Abstract]
Indoor air quality has a critical impact on human health due to the presence of indoor air pollutants. Small home appliances equipped with heating coils and brushed motors generate ultrafine particles that directly attack users. In this study, the physicochemical characteristics of ultrafine particles (UFPs) emitted from selected appliances sold in South Korea were investigated using online real-time aerosol instruments and offline analyses. The brushed motor-embedded hair dryers mostly produced nucleation-mode UFPs, while air fryers and toasters generated Aitken mode-size UFPs. In contrast, the two brushless motor-equipped hair dryers showed the lowest particle emission (at least 1.4 times lower) than others. Among the UFPs, distinct particles composed of copper, silver, or titanium were observed. Carbon-containing particles were also found, which could be produced by the incomplete combustion of organics in the inddor air. The MPPD model simulation showed that the mass per unit area of deposited particles in lungs were higher in children than adults. This implies that long exposure to UFPs emitted from small home appliances can cause unexpected health impacts and diseases more in children than adults. Regulation and test protocols for UFP emissions from home appliances are required to achieve clean indoor air quality for human health.
- Authors (Pusan National University): Geun-wu Ryu, Jeongbeen Kim, Changhyuk Kim (School of Civil and Environmental Engineering, Department of Environmental Engineering)
- Title of original paper: Physicochemical characteristics and health impacts of ultrafine particles emitted from small home appliances equipped with heating coils and brushed motors
- Journal: Journal of Hazardous Materials
- Web link: https://doi.org/10.1016/j.jhazmat.2025.139796
- Contact e-mail: changhyuk.kim@pusan.ac.kr
-
해양 메탄 관리 과학적 근거 제시, 기후변화 연구 도움
해양학과/_UPLOAD/IMAGE/Board/68/2026/1/OcTeE3jX4TttdHqe.JPG해양학과정동주 교수팀 "심해 메탄, 대기까지 못 간다…지구온난화 영향 없어"해양 메탄 관리 과학적 근거 제시, 기후변화 연구 도움
지구의 기후변화에 큰 역할을 하는 ‘메탄’은 해양 중층 해저 탄화수소 누출(hydrocarbon seeps)로부터 다량 용출된 뒤 대기로 유입돼 지구온난화를 가속한다는 것이 기존의 대표적인 가설이었다. 하지만 이를 뒤집는 흥미로운 연구 결과가 나왔다.해양학과 정동주 교수(G-LAMP 전임교원)가 미국 로체스터대 연구진과의 국제공동연구를 통해, 수심 500m보다 깊은 가스하이드레이트(gas hydrate, 일명 ‘불타는 얼음’) 및 해저 탄화수소 누출 지역에서 발생하는 메탄은 대부분 바닷물에 녹거나 미생물에 의해 소비돼 대기 중 메탄 증가에는 사실상 영향을 주지 않는다는 새로운 연구 결과를 발표했다. 이번 연구는 기후변화 예측의 정확도를 높이고 해양 메탄 관리·정책 수립에 과학적 근거를 제공할 전망이다.
메탄(methane)은 이산화탄소보다 약 80배나 강한 온실효과를 가진 기체로, 최근 그 사용량이 급격히 늘고 있다. 또한 지구온난화로 자연에서 방출되는 메탄이 대기로 유입되는 속도가 빨라지면서, 메탄은 향후 기후변화의 향방을 좌우할 핵심 요인으로 더욱 주목받고 있다.
해저 탄화수소 자연 누출(seep)은 가스하이드레이트 분해나 지하 퇴적층에서 생성된 다량의 메탄이 기포(bubble)나 용해된 형태로 바닷물에 흘러나오는 현상을 말한다. 이러한 메탄 유출 지점은 전 세계 바다의 얕은 연안부터 깊은 심해까지 다양한 수심대 해저에 걸쳐 광범위하게 분포한다.
특히 바다는 지구상에서 메탄을 가장 많이 저장하고 있는 장소로 알려져 있으며, 지구온난화로 해수 온도가 상승함에 따라 해양 중층에서의 메탄 방출도 더욱 활발해질 것으로 우려된다.
지금까지 과학계의 일반적인 가설은 ‘해저에서 분출된 메탄이 대기까지 도달하면 지구온난화를 가속화시킨다’는 것이었다. 이는 메탄 유출 → 지구온난화 → 추가 메탄 유출로 이어지는 이른바 ‘메탄-기후변화 양성 피드백 고리(positive feedback loop)’, 즉 결과가 원인을 더욱 증폭시키는 악순환을 의미한다.
실제로 현재 진행 중인 지구온난화로 해수 온도가 올라가면 해저의 가스하이드레이트가 붕괴되고 메탄이 해수와 대기로 대량 유출되며, 이로 인해 지구온난화가 한층 가속돼 다시 해수 온도가 상승하는 식의 폭주 메커니즘이 우려돼 왔다. 그러나 이를 뒷받침하는 실험적 증거는 부족했고, 해수에 용해된 메탄에 대한 연구도 거의 전무한 실정이었다.

【걸프만 지역에서의 메탄의 분포】
A) 걸프만 연구지역 및 해저에 존재하는 자연발생적 메탄 유출지 (natural seeps)
B) 연구지역 메탄 농도와 방사성 탄소의 수직 분포
C) 걸프만 지역 해수 내 메탄의 방사성 탄소의 분포 예상도
이러한 배경에서 부산대 연구팀은 메탄에 포함된 방사성 탄소 동위원소(14C-CH₄)를 정밀 분석해, 바다 표면부터 심층에 이르는 해수 내 메탄의 기원을 밝혀내는 연구를 수행했다. 연구팀은 미국 멕시코만 지역의 해저 가스하이드레이트 분해 및 자연 탄화수소 누출지에서 발생한 메탄을 조사한 결과, 심해에서 방출된 메탄이 바닷물 속으로는 대량 녹아들지만 끝내 바다 표면까지 도달하지 못한다는 사실을 실험적으로 최초 확인했다.
이 결과는 정동주 교수가 대서양과 태평양 심해를 대상으로 수행한 선행 연구(2022, 『Nature Geoscience』 게재)와도 맥을 같이한다. 당시 연구에서도 수심 500m보다 깊은 바다에서 분출된 해저 메탄은 거의 모두 바닷속에 머무르고 대기로의 직접 유입량은 극히 미미함이 밝혀졌다. 다시 말해, 500m 이상 깊은 해역의 해저 메탄은 우려됐던 ‘메탄-지구온난화-기후변화’ 양성 피드백 고리에 유의미한 영향을 주지 못한다는 점을 이번 연구가 실험적으로 입증한 것이다.
한편 연구진은 상대적으로 얕은 수심의 자연발생 유출지(natural seeps)에서 방출되는 메탄은 해수 표층 및 대기로 실제 유입됨을 함께 확인했다.

【걸프만 수심 500m에서 해수 내 메탄 추출 시스템】
이번 연구를 주도한 정동주 교수(제1저자 겸 교신저자)는 “메탄은 기후변화에 직접적인 영향을 주는 기체임에도 불구하고, 관련 연구가 전 세계적으로 아직 많이 부족하고, 한국에서는 손에 꼽을 정도로 적은 실정”이라며 “메탄 사용량 증가와 이산화탄소보다 훨씬 강력한 메탄의 온실효과를 고려할 때 메탄 연구는 앞으로의 기후변화 연구에서 핵심적인 분야가 될 것”이라고 강조했다.
정 교수는 이어 “한국은 동아시아에서 대기 중 메탄 농도가 가장 빠르게 높아지는 나라”라며 “강·호수·연안습지·갯벌·만 등 다양한 자연환경과 간척지·양식장·농경지 같은 인공환경이 잘 발달해 메탄 발생에 매우 유리한 조건을 갖추고 있다”고 설명했다. 그럼에도 불구하고 국내 메탄 관련 연구는 아직 미흡한 수준이라며, “전 지구적인 온실가스 감축 노력에 앞서 다양한 메탄 분야의 기초 및 응용 연구가 선행돼야 하고, 이를 위해 정부의 적극적인 지원이 매우 중요하다”고 덧붙였다.
이번 연구에서는 메탄 내 방사성 탄소를 정밀 측정하는 새로운 분석 기술을 적용했다. 이 기술은 측정 과정에서 발생하는 오차를 크게 줄여 데이터 정확도를 획기적으로 높여주는데, 현재 전 세계에서 이 기술을 실제 연구에 활용하고 있는 팀은 정 교수 연구팀이 유일하다. 정 교수는 “이 분야만큼은 한국이 세계를 선도할 잠재력이 크다”고 말했다.
정동주 교수는 또 “해저 메탄의 직접적인 대기 유입은 매우 미미하지만, 해수 속에서 미생물에 의해 메탄이 산화되면 이산화탄소로 바뀌어 해양의 이산화탄소 농도를 높이고 결국 바다가 대기에서 이산화탄소를 흡수하는 능력을 약화시킬 수 있다”고 짚었다.
정 교수는 “이번 연구는 걸프만 해역에 국한된 결과지만, 앞으로 극지방이나 우리나라 동해 등 다양한 해역으로 연구를 확대한다면 해양 메탄의 대기 유입에 대한 훨씬 정교한 이해를 제공하게 될 것이고, 전 지구적 기후변화 연구에도 크게 기여할 것으로 기대한다“고 전망했다.
기존 이론에 도전하는 새로운 관점을 제시한 이번 연구는 부산대와 미국 로체스터대학교의 존 케슬러(John D. Kessler) 교수와 토마스 웨버(Thomas Weber) 교수와의 국제공동연구로 수행됐으며, 관련 논문은 국제 과학저널 『Communications Earth and Environment』 2025년 12월 15일자에 게재됐다.
- 논문 제목: Deep Gulf of Mexico seeps are not a significant source of methane to the atmosphere (맥시코만 심해 메탄은 대기 메탄의 주요 공급원이 아니다)
- 논문 링크: https://www.nature.com/articles/s43247-025-03027-0
* 상단 사진: 정동주(왼쪽 네 번째) 교수와 연구실 학생들.
[Abstract]
Hydrocarbon seeps release fossil methane into the marine environment, but emission of this seep-derived methane to the atmosphere is challenging to constrain. Here, we measure the concentration and radiocarbon content of dissolved methane in seawater above seeps in the northern Gulf of Mexico that have previously been linked to considerable atmospheric emissions. In bottom waters above the seeps, methane radiocarbon content is close to zero, confirming the release of fossil seep methane. However, radiocarbon signatures approach modern values at shallower depths, indicating that only ~21% of the methane in surface waters is sourced from seeps. We observe a mid-depth methane concentration maximum and radiocarbon minimum at ~200m below the surface, but this likely reflects lateral advection of fossil methane from shallower seep fields. Our findings are consistent with previous radiocarbon fingerprinting in coastal regions, and suggest that seeps deeper than ~400m are not a major contributor to atmospheric methane emissions.
- Author (Pusan National University): DongJoo Joung (Deapartment of Oceanography, G-LAMP)
- Title of original paper: Deep Gulf of Mexico seeps are not a significant source of methane to the atmosphere
- Journal: Communications Earth and Environment
- Web link: https://www.nature.com/articles/s43247-025-03027-0
- Contact e-mail: dongjoo.joung@usan.ac.kr
-
지구온난화에 따른 해빙 감소로, 향후 극지 해양의 수평 교란 강화될 것
부산대 IBS 기후물리 연구단/_UPLOAD/IMAGE/Board/68/2026/1/tBK5s4zqcKSXR0fd.JPG부산대 IBS 기후물리 연구단이준이 교수팀 "지구온난화로 녹는 해빙, 바다의 역동성 강화시킨다"지구온난화에 따른 해빙 감소로, 향후 극지 해양의 수평 교란 강화될 것
지구온난화의 가속화로 극지방의 해빙이 전례 없는 속도로 빠르게 녹고 있다. 이에 따라 해류의 흐름이 불안정해지며 앞으로 극지 바다가 더욱 거세게 요동치고, 불안정해질 것이라는 분석이 제기됐다.부산대 IBS(기초과학연구원) 기후물리 연구단 이준이 교수 연구팀은 초고해상도 지구 시스템 모델 시뮬레이션을 통해 인간에 의한 온난화가 해빙을 빠르게 녹여, 바다의 ‘중규모 수평 교란 현상’이 더욱 강화될 것이라고 밝혔다.
‘중규모 수평 교란(mesoscale horizontal stirring)’은 바람, 해류, 그리고 바다 속 소용돌이 등이 만들어 내는 거대한 물결 섞임 현상이다. 수십에서 수백 킬로미터 규모로 바닷물이 수평 방향으로 휘저어지며, 열과 영양분이 퍼지고 플랑크톤이나 어란·유충, 미세 플라스틱과 같은 오염 물질 확산에도 영향을 미친다.
하지만 이러한 극지 해양의 변화를 직접 관측하는 일은 쉽지 않다. 특히 지구온난화가 극지역 소규모 해류와 해양 생태계에 미치는 영향을 정량적으로 규명하는 일은 여전히 어려운 과제로 남아 있다. 지리적 한계로 관측이 제한되고, 위성 관측 자료로는 중규모 해양 과정을 정밀하게 파악하기 어렵기 때문이다.
이에 연구팀은 IBS 슈퍼컴퓨터 알레프(Aleph)를 활용해 초고해상도 기후모델(CESM-UHR) CESM-UHR*을 이용한 시뮬레이션을 수행했다. 대기 중 이산화탄소 농도를 현재 수준, 2배, 4배로 설정해 비교한 결과, 이산화탄소 농도가 높아질수록 북극과 남극 연안의 바다가 더욱 거세게 요동쳤다. 온난화가 심화될수록 중규모 수평 교란이 뚜렷하게 증가하는 것이다.
* CESM-UHR: 완전 결합 기후모델로 대기–해빙–해양 구성 요소를 통합하고, 대기 0.25⁰, 해양 0.1⁰의 수평 해상도를 통해 기후시스템 내 상호작용을 보다 현실적으로 재현한다.
연구진은 이 현상을 정량화하기 위해 ‘유한 크기 리아푸노프 지수(Finite-Size Lyapunov Exponent, FSLE)’*를 활용했다. 이 지수는 가까운 두 유체 입자가 얼마나 빠르게 분리되는지를 보여주며, 값이 클수록 교란이 강화되며 해수의 움직임이 더 활발해지는 것을 의미한다.
* 유한 크기 리아프노프 지수[finite-size Lyapunov exponents(FSLE)]: 유체의 입자 간 분리 속도를 정량화하는 지표로 두 입자가 일정 거리에서 출발해 더 큰 거리로 벌어지는 데 걸리는 시간을 이용해 계산됨. 값이 커질수록 공간적으로 더 빠르게 분리됨을 의미함.
시뮬레이션 결과, 지구온난화로 인한 해빙의 급격한 감소가 미래 북극해 및 남극 연안 해역의 해류와 난류를 강화하며, 바닷물의 수평 교란을 가속화하는 것으로 나타났다.

【대기 중 이산화탄소 현재 농도(왼쪽) 및 4배증(오른쪽) 조건에서의 북극해 3월 중규모 수평 교란 비교(위 그림)와 남극해 9월 중규모 수평 교란 비교(아래 그림)】
중규모 수평 교란은 유한 크기 리아프코프 지수(FSLE)를 이용해 정량화했다. FSLE가 높은 값을 가질수록(밝을수록) 수평 교란이 더 강하게 나타남을 의미한다.
그림 출처: IBS 기후물리 연구단 학생 연구원 이규석 (그림의 기초가 되는 지도는 NASA Visible Earth (https://visibleearth.nasa.gov/)에서 차용)
더불어, 연구진은 북극과 남극에서 교란이 강화되는 원인이 서로 다르다는 점을 밝혀냈다. 북극해에서는 해빙이 줄면서 바람이 해수를 더 강하게 밀어 표층 순환류와 난류를 강화시키는 반면, 남극 연안 해역에서는 녹은 해빙에 의한 담수 유입이 해수의 밀도 차이를 키워 해류 세기와 교란을 강화시키는 것으로 나타났다.
제1저자인 이규석 학생 연구원은 “대륙에 둘러싸인 북극해와 남극 연안 해역의 대조적인 지리적 구조 차이는 해수의 수평 교란 변화를 결정하는 역학 과정에서 근본적인 차이를 가져온다. 그럼에도 지구온난화가 지속될 경우, 두 해역 모두에서 수평 교란이 크게 강화될 것으로 예상된다”라고 설명했다.교신저자인 이준이 IBS 기후물리 연구단 및 기후과학연구소 교수는 “미래 극지 해양 수평교란의 증가는 어란·유충의 생존을 포함해 극지 해양 생태계에 큰 영향을 미칠 수 있다”라고 덧붙였다.
악셀 팀머만 IBS 기후물리 연구단장은 “현재, 우리 연구단에서는 기후와 생명의 상호작용을 보다 효과적으로 통합하는 차세대 지구 시스템 모델을 개발 중이다. 이를 통해, 극지 생태계가 지구온난화에 어떻게 반응하는지에 대한 이해를 높일 수 있을 것”이라며 향후 연구계획을 밝혔다.
이번 연구 결과는 2025년 11월 5일 국제학술지 『Nature Climate Change』에 게재됐다.
- 논문 제목: Future mesoscale horizontal stirring in polar oceans intensified by sea ice decline (해빙 감소로 인해 극지방 해양의 미래 중규모 수평 교란 심화 전망)
- 논문 링크: https://doi.org/10.1038/s41558-025-02471-2
* 상단 연구진 사진: 왼쪽부터 이준이 교수, 이규석 학생 연구원.
[Abstract]
More Polar Ocean Turbulence due to Planetary Warming
A study published in the journal Nature Climate Change by an international team of scientists, from the IBS Center for Climate Physics (ICCP) at Pusan National University in South Korea, presents new evidence that ocean turbulence and a process known as “horizontal stirring” will increase dramatically in the Arctic and Southern Oceans due to human-induced Global Warming and decreasing sea ice coverage.
“Shaken, not stirred” - it is widely known how James Bond prefers his Martinis. Stirring works by stretching a fluid into thin streaks, which eventually helps to create turbulence, contributing to the mixing of water properties. In the ocean, a similar stirring process of seawater occurs due to the action of winds and other sources of energy. When it happens horizontally over scales of tens to hundreds of kilometers, it is referred to as mesoscale horizontal stirring (MHS). MHS transports and redistributes heat and nutrients, thereby determining the distribution of plankton in the upper ocean. Moreover, the stretching, rotation, and spatial separation of nearby fluid parcels over time also control the dispersal of fish eggs and larvae, but also of pollutants, such as microplastics.
Due to the remoteness of polar regions, it has remained challenging to study the impact of global warming on small-scale ocean currents and marine ecosystems using ship-based observations and satellite data. Instead, climate scientists have relied heavily on climate computer models. However, the current generation of such models lacks the spatial resolution to resolve small-scale ocean processes relevant to MHS and the production of turbulence and horizontal mixing.
To overcome this shortcoming, the South Korean research team analyzed results from ultra-high-resolution simulations conducted with the Community Earth System Model version 1.2.2 (CESM-UHR), on the Aleph supercomputer at the Institute for Basic Science in Daejeon. This fully-coupled model integrates atmosphere, sea ice, and ocean components to realistically represent their interactions within the climate system, using a horizontal resolution of 0.25° for the atmosphere and 0.1° for the ocean. The team focused on simulations under present-day (PD), CO2 doubling (2xCO2), and quadrupling (4xCO2) conditions to investigate how MHS responds to human-induced warming.
To characterize the stretching of fluids into elongated filament-like structures (Fig. 1), the research team employed a technique known as finite-size Lyapunov exponents (FSLE), which tracks how quickly neighboring fluid parcels separate over time due to mesoscale ocean eddies (swirling currents with scales of tens to hundreds of kilometers), meandering flows, and ocean fronts. Using daily data from 10 years of simulation, the computationally demanding FSLE calculations show a pronounced future intensification of MHS across the Arctic Ocean and along the Antarctic coastal region (Fig. 1,2), which can be attributed primarily to the dramatic decline in sea ice in a warming world. The researchers found that the mechanisms linking sea ice loss to enhanced MHS differed between the two regions.
In the Arctic Ocean, the disappearance of sea ice increases the mechanical energy input into the ocean. Uninterrupted by sea ice, a clockwise wind forcing can strengthen both the mean ocean flow and enhance the generation of upper ocean eddies, ultimately leading to intensified MHS (Fig. 1) and turbulence.
By contrast, in the Antarctic coastal region, the projected future strengthening of MHS around Antarctica arises from near-shore freshening due to sea ice decline, which enhances the north-south density gradient. This, in turn, reinforces the mean ocean currents, such as the Antarctic Slope Current, enhancing eddy activity and MHS (Fig. 2). Given that such intensification of MHS is expected to induce major changes in ocean ecosystems as well as in the dispersal of marine pollutants, further research is urgently needed.
“The contrast between the Arctic Ocean, which is enclosed by surrounding continents, and the Southern Ocean, where the continent is encircled by ocean, creates different physical conditions for ocean stirring. But the outcome for ocean stirring under warming is quite similar,” said lead author Gyuseok Yi, a doctoral researcher at the ICCP and Pusan National University.
“Horizontal stirring is a crucial factor for fish larval transport across the ocean. For moderate values, this process connects populations and habitats geographically, increasing their genetic exchange. However, for increasing stirring in the future, larvae can be transported into unsuitable areas where they may not survive,” remarks Prof. June-Yi Lee from the ICCP and co-corresponding author of the study.
Understanding the ecological implications of the author’s main findings requires additional earth system modeling experiments at high spatial resolution, including computer models of plankton and fish.
“Currently, at the IBS Center for Climate Physics in South Korea, we are developing a new generation of earth system models that better integrates the interactions between climate and life. This will deepen our understanding of how polar ecosystems respond to Global Warming”, said Prof. Axel Timmermann, co-author of the study and director of the ICCP.
- Authors (Pusan National University): Gyuseok Yi, June-Yi Lee (Institute for Basic Science Center for Climate Physics)
- Title of original paper: Future mesoscale horizontal stirring in polar oceans intensified by sea ice decline
- Journal: Nature Climate Change
- Web link: https://doi.org/10.1038/s41558-025-02471-2
- Contact e-mail: juneyi@pusan.ac.kr